参考基因组下载方式汇总
基因组学数据分析的第一步通常是搭建分析流程,而分析流程的第一步通常是下载分析的参考数据,这里我总结下载fasta的几种方式。
UCSC goldenPath
以hg19为例:
http://hgdownload.cse.ucsc.edu/goldenPath/hg19/bigZips/chromFa.tar.gz
解压之后是按染色体分开的多个文件,需要自己合并再建索引。
但是UCSC上可以很方便的获取一段基因组的序列,如:
http://genome.ucsc.edu/cgi-bin/das/hg38/dna?segment=chr17:7676091,7676196
Ensembl biomart
以hg19为例:
http://ftp.ensembl.org/pub/release-56/fasta/homo_sapiens/dna/
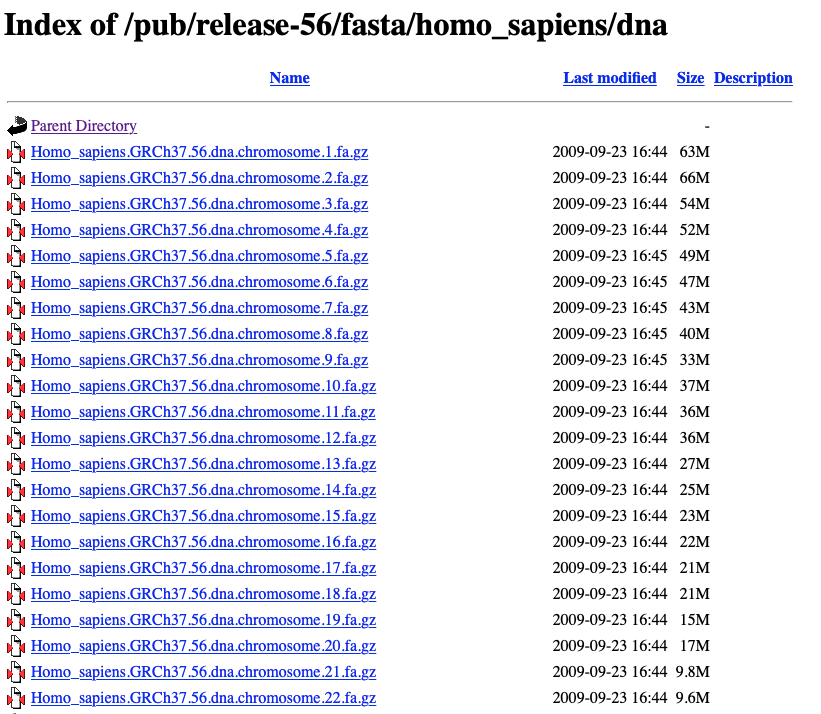
也是只有单个染色体的文件
GO GET DATA (GGD)
GGD是一个参考数据的管理软件,收集整理了包括人和小鼠的常见的参考数据。
安装
1 | |
搜索
1 | |
或者直接在他们的数据集里面找:https://gogetdata.github.io/recipes.html#recipes
下载,以hg19为例
1 | |
Google life science
Google life science是谷歌云的生物数据业务,其将一部分参考数据放在了Google storage上,地址在这里,不过只有人的数据,而且比较乱,更多的是DNAseq分析要用的数据,比如GATK variant call。
下载需要用Google storage的命令行工具 gsutli
安装:
1 | |
下载示例:
1 | |
AWS iGenomes
https://ewels.github.io/AWS-iGenomes/
这是大佬Phil Ewels开发的,参考数据除了有fasta以外还有GTF和BED文件,甚至还可以直接下载常见比对软件的索引文件,这样就可以节省大把的建索引的时间。而且文件放在AWS上,下载很方便,速度很快。
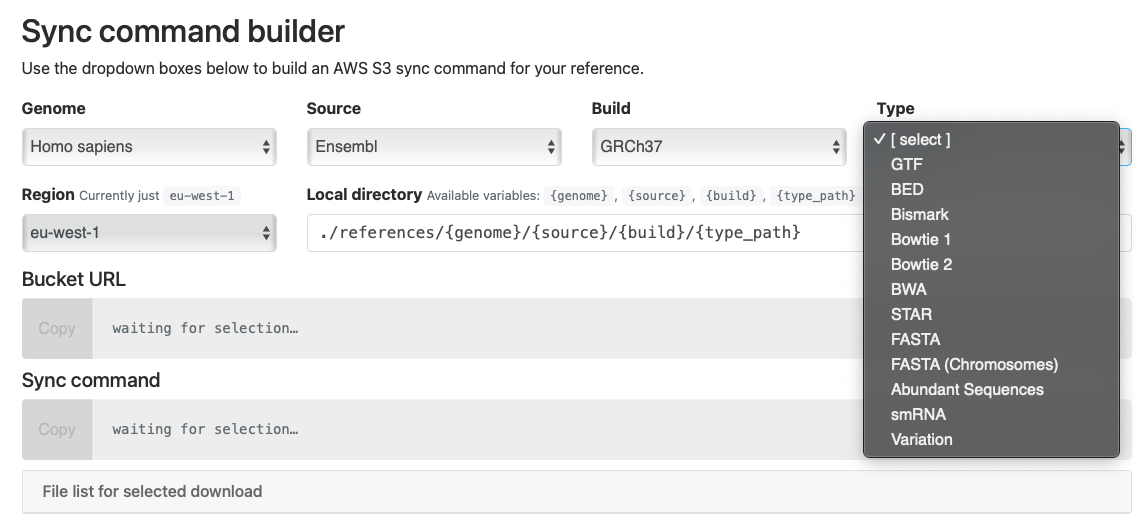
安装awscli
1 | |
下载示例:
1 | |
总结
这些方式各有各的好处,总结一下就是:
如果是用来bwa/bowtie等比对软件,可以直接在AWS iGenomes下载,又快又好;
如果是要用来跑GATK variant calling流程,必须要用Google life science上面GATK配套的数据;
如果是想获取一段DNA序列,用UCSC和biomart都可以。